【机器学习】异常检测文献阅读:IsolationForest拓展
本文梳理基于 Isolation Forest(IF)的若干拓展算法
关于 IF 的相关知识我在之前的文章【机器学习】异常检测文献阅读:关键算法篇 中进行了梳理
Extended Isolation Forest
Rotated Isolation Forest
Fuzzy Set-Based Isolation Forest
Efficient Anomaly Detection by Isolation Using Nearest Neighbour Ensemble
1 Extended Isolation Forest(EIF)
1.1 简介
原有的算法 IF 利用了异常数据总是少而与众不同的特点,通过一次次的随机分割数据建立隔离森林来分离出异常值,足够直观且高效。
然而分割数据的方式是每次分割是个数据的某一单一维度上进行的,所以每次运行的结果总会出现一定的偏差。

通过 正态数据分布的异常分数图 可以看出,异常分数总是随着到某一数据中心的距离的增加而增加,IF单纯的从某一维度对数据切割,总会存在偏差。

同样对于两簇正态分布的数据,使用 IF 不断选取单一维度进行切割,不难发现在数据右上角与左下角也会产生异常分数较低的簇(gohst clusters)(实际上这些位置的点应该为异常点)。

对于正弦波形也是同理。

试想我们如果可以斜着切割数据,那么就可以有效弥补这一不足。
在 EIF 中,对 IF 中的数据分割方式进行了改进,来弥补原有算法的缺陷。
改进的方法中,EIF 使用了一个拥有随机斜度(slopes)的超平面来对数据进行切割,该超平面不与坐标轴平行。

1.2 算法描述
Isolation Forest 分支切口(branchs cuts)的信息:
- 一个随机选取的特征或坐标
- 一个来自于该特征的随机值
Extended Isolation Forest 分支切口信息:
- 一个分支切口的随机斜度(slope)
- 一个在数据集可用值范围内随机选择的截距(intercept)
二维空间如此,高维空间(N 维)也是同理,只不过分支切口变味了一个 N -1 维的超平面
这些超平面同样被一个随机正交向量和一个随机截距点所确定,如二维情况类似
1.3 算法实现



1.4 测试





2 Fuzzy Set-Based Isolation Forest(FSBIS)
2.1 简介
文章在原有算法 IF 的基础上,提出通过对记录的属性的隶属度(membership)进行划分从而构建森林。
其中,隶属度是根据到样本中心(middle of the cluster)的距离(也就是属性的平均值)来确定的。
优点:
- 直观
- 返回异常值的隶属度几乎为零
- 以返回异常程度的方式构造的函数不需要使用复杂的标准化公式
- 运行时间并不比经典 IF 多,在某些情况下甚至短于经典 IF
2.2 算法描述
对于经典 IF 相对复杂的建树过程,FSBIS 提出了如下改进
在经典 IF 的第一阶段中,我们会从过滤后的簇(cluster)中所选择的属性的所有值中确定平均值 m,这个值会被存在相应的节点上
1
TODO 平均值m的具体确定方法?
we always determine the average value of m from all the values of the chosen attribute located in the filtered cluster.
通过公式计算出已经构建好的簇的隶属度
最终,异常分数是每个节点所有隶属度之和
2.3 算法实例

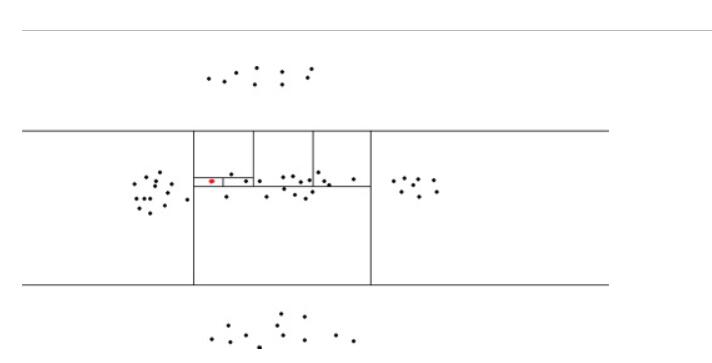
1 | 如上图所示,我们要计算异常分数的点是 (x, y) = (0.7, 0.6) |
构建的二叉树如下所示:

2.4 算法实现




3 Efficient Anomaly Detection by Isolation Using Nearest Neighbour Ensemble
Advantage:
- Compared with some nearest neighbour-based method, the proposed method has linear time complexity and constant space complexity.
- Compared with tree-based isolation method iForest, the proposed isolation method overcomes three weaknesses of iForest
- Its inability to detect local anomalies
- anomalies with a low number of relevant attributes
- anomalies that are surrounded by normal instances
Two important challenging:
- the requirement of low computational cost
- the susceptibility to issues in high-dimensional datasets
How to operate:
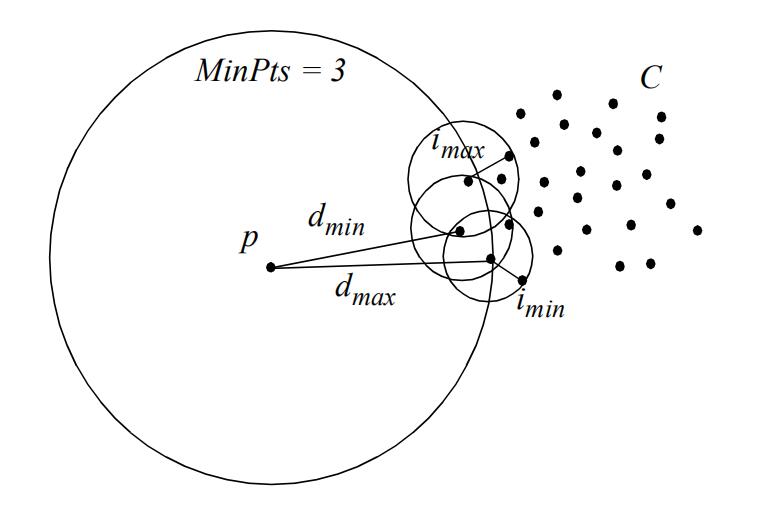
- it partitions the data space into regions using a subsample and determines an isolation score for each region
- each region is defined with a centre represented by an instance from the subsample; and its boundary is defined by the distance to the nearest neighbour of the instance at the centre.
The intuition behind iNNE:
- an anomaly can be expected to be far from its nearest neighbour
Some definations:
Definition 1
Definition 2
Definition 3


Defination 4
Where β is a user defined threshold.
Defination 5
Defination 6
Defination 7
Defination 8
$$\begin{pmatrix} 1/\sqrt{2} & 1/\sqrt{2} \ -1/\sqrt{2} & 1/\sqrt{2} \end{pmatrix} \begin{pmatrix} 1 & 2 & 3 \ 1 & 2 & 3 \end{pmatrix} = \begin{pmatrix} 2/\sqrt{2} & 4/\sqrt{2} & 6/\sqrt{2} \ 0 & 0 & 0 \end{pmatrix}$$
$$\begin{pmatrix} 1/\sqrt{2} & 1/\sqrt{2} \ -1/\sqrt{2} & 1/\sqrt{2} \end{pmatrix} \begin{pmatrix} 3 \ 2 \end{pmatrix} = \begin{pmatrix} 5/\sqrt{2} \ -1/\sqrt{2} \end{pmatrix}$$
$$\begin{pmatrix} p_1 \ p_2 \ \vdots \ p_R \end{pmatrix} \begin{pmatrix} a_1 & a_2 & \cdots & a_M \end{pmatrix} = \begin{pmatrix} p_1a_1 & p_1a_2 & \cdots & p_1a_M \ p_2a_1 & p_2a_2 & \cdots & p_2a_M \ \vdots & \vdots & \ddots & \vdots \ p_Ra_1 & p_Ra_2 & \cdots & p_Ra_M \end{pmatrix}$$
$$\begin{pmatrix} 1 & 1 & 2 & 4 & 2 \ 1 & 3 & 3 & 4 & 4 \end{pmatrix}$$
$$\begin{pmatrix} -1 & -1 & 0 & 2 & 0 \ -2 & 0 & 0 & 1 & 1 \end{pmatrix}$$
$$Var(a)=\frac{1}{m}\sum_{i=1}^m{(a_i-\mu)^2}$$
$$Cov(a,b)=\frac{1}{m}\sum_{i=1}^m{a_ib_i}$$
$$X=\begin{pmatrix} a_1 & a_2 & \cdots & a_m \ b_1 & b_2 & \cdots & b_m \end{pmatrix}$$
$$\frac{1}{m}XX^\mathsf{T}=\begin{pmatrix} \frac{1}{m}\sum_{i=1}^m{a_i^2} & \frac{1}{m}\sum_{i=1}^m{a_ib_i} \ \frac{1}{m}\sum_{i=1}^m{a_ib_i} & \frac{1}{m}\sum_{i=1}^m{b_i^2} \end{pmatrix}$$
$$\begin{array}{l l l} D & = & \frac{1}{m}YY^\mathsf{T} \ & = & \frac{1}{m}(PX)(PX)^\mathsf{T} \ & = & \frac{1}{m}PXX^\mathsf{T}P^\mathsf{T} \ & = & P(\frac{1}{m}XX^\mathsf{T})P^\mathsf{T} \ & = & PCP^\mathsf{T} \end{array}$$
$$E=\begin{pmatrix} e_1 & e_2 & \cdots & e_n \end{pmatrix}$$
$$E^\mathsf{T}CE=\Lambda=\begin{pmatrix} \lambda_1 & & & \ & \lambda_2 & & \ & & \ddots & \ & & & \lambda_n \end{pmatrix}$$
$$P=E^\mathsf{T}$$